[1] FALSERedes egocéntricas
Esta versión del capítulo fue traducida de manera automática utilizando IA. El capítulo aún no ha sido revisado por un humano.
En el análisis de redes sociales egocéntricas (ESNA, para nuestro libro,) en lugar de tratar con una sola red, tenemos tantas redes como participantes en el estudio. Los egos–los sujetos principales del estudio–se analizan desde la perspectiva de su red social local. Para una vista más extendida de ESNA, revisa “Análisis de redes egocéntricas con R” de Raffaele Vacca.
En este capítulo, muestro cómo trabajar con un tipo particular de datos ESNA: información generada por la herramienta Network Canvas. Puedes descargar un archivo ZIP “artificial” que contiene las salidas de un proyecto de Network Canvas aquí1. Asumimos que el archivo ZIP fue extraído a la carpeta data-raw/egonets. Puedes proceder y extraer el ZIP por punto y clic o usar el siguiente código R para automatizar el proceso:
unzip(
zipfile = "data-raw/networkCanvasExport-fake.zip",
exdir = "data-raw/egonets"
)Esto extraerá todos los archivos en networkCanvasExport-fake.zip a la subcarpeta egonets. Echemos un vistazo a los primeros archivos:
head(list.files(path = "data-raw/egonets"))
## [1] "I_-59190_BRB9111_attributeList_Person.csv"
## [2] "I_-59190_BRB9111_edgeList_Knows.csv"
## [3] "I_-59190_BRB9111_ego.csv"
## [4] "I_-59190_BRB9111.graphml"
## [5] "I-100BB_00B95-90_attributeList_Person.csv"
## [6] "I-100BB_00B95-90_edgeList_Knows.csv"Como puedes ver, para cada ego en el conjunto de datos, hay cuatro archivos:
...attributeList_Person.csv: Atributos de los alters....edgeList_Knows.csv: Lista de enlaces indicando los vínculos entre los alters....ego.csv: Información sobre los egos....graphml: Y un archivographmlque contiene las redes egocéntricas.
Las siguientes secciones ilustrarán, archivo por archivo, cómo leer la información en R, aplicar cualquier procesamiento requerido, y almacenar la información para uso posterior. Comenzamos con los archivos graphml.
Archivos de red (graphml)
Los archivos graphml pueden leerse directamente con la función read_graph de igraph. La clave es aprovechar las listas de R para evitar escribir una y otra vez el mismo bloque de código, y, en su lugar, manejar los datos a través de listas.
Al igual que cualquier función de lectura de datos, la función read_graph requiere una ruta de archivo al archivo de red. La función que usaremos para listar los archivos requeridos es list.files():
# Comenzamos cargando igraph
library(igraph)
# Listando todos los archivos graphml
graph_files <- list.files(
path = "data-raw/egonets", # ¿Dónde están estos archivos?
pattern = "*.graphml", # Especificar un patrón para solo listar graphml
full.names = TRUE # Y nos aseguramos de usar el nombre completo
# (ruta.) De lo contrario, solo obtendríamos nombres.
)
# Echando un vistazo a los primeros tres archivos que obtuvimos
graph_files[1:3]
## [1] "data-raw/egonets/I_-59190_BRB9111.graphml"
## [2] "data-raw/egonets/I-100BB_00B95-90.graphml"
## [3] "data-raw/egonets/I-1BB79950-0-7.graphml"
# Aplicando read_graph de igraph
graphs <- lapply(
X = graph_files, # Lista de archivos a leer
FUN = read_graph, # La función a aplicar
format = "graphml" # Argumento pasado a read_graph
)Si la operación tuvo éxito, el bloque de código anterior debería generar una lista de objetos igraph llamada graphs. Echemos un vistazo a los primeros dos:
graphs[[1]]
## IGRAPH 8d9ee25 U--- 12 25 --
## + attr: age (v/n), healthy_diet (v/n), gender_1 (v/l), eat_with_2
## | (v/l), id (v/c)
## + edges from 8d9ee25:
## [1] 1-- 3 1-- 2 1-- 6 1-- 5 1-- 4 1-- 8 1--11 1--10 2-- 3 3-- 7 3-- 4 3-- 5
## [13] 3-- 6 2-- 7 2-- 4 2-- 5 2-- 6 5-- 6 6--10 7-- 9 4-- 5 5-- 7 4--11 6-- 7
## [25] 4-- 7
graphs[[2]]
## IGRAPH 6908c8e U--- 16 47 --
## + attr: age (v/n), healthy_diet (v/n), gender_1 (v/l), eat_with_2
## | (v/l), id (v/c)
## + edges from 6908c8e:
## [1] 7--13 1-- 5 1-- 6 1-- 4 1-- 2 7--15 1-- 3 11--13 1--10 1--16
## [11] 4-- 6 2-- 6 6-- 7 1--11 11--15 6-- 9 6-- 8 3-- 9 5--15 4-- 5
## [21] 2-- 5 5-- 8 5-- 7 5--10 3-- 5 6--14 12--13 6--13 3--13 2-- 3
## [31] 3-- 4 3--16 3--11 10--14 7--14 2-- 4 2--10 2--15 10--12 4-- 7
## [41] 6--10 5--11 9--10 1-- 9 1--12 3--12 4--14Como siempre, una de las primeras cosas que hacemos con redes es visualizarlas. Usaremos el paquete de R netplot (de un servidor) para dibujar las figuras:
library(netplot)
library(gridExtra)
# El diseño del grafo es aleatorio
set.seed(1231)
# grid.arrange permite poner múltiples gráficos netplot en la misma página
grid.arrange(
nplot(graphs[[1]]),
nplot(graphs[[2]]),
nplot(graphs[[3]]),
nplot(graphs[[4]]),
ncol = 2, nrow = 2
)
¡Excelente! Dado que los nodos en nuestra red tienen características, podemos agregar un poco de color. Usaremos la variable eat_with_2, codificada como TRUE o FALSE. Los colores de los vértices pueden especificarse usando el argumento vertex.color de la función nplot. En nuestro caso, especificaremos colores pasando un vector con longitud igual al número de nodos en el grafo. Además, dado que haremos esto múltiples veces, vale la pena escribir una función:
# Una función para colorear por la variable come con
color_it <- function(net) {
# Codificando eat_with_2 para ser 1 (FALSE) o 2 (TRUE)
eatswith <- V(net)$eat_with_2
# Subconjuntando el color
ifelse(eatswith, "purple", "darkgreen")
}Esta función toma dos argumentos: una red y un vector de dos colores. Los atributos de vértice en igraph pueden accederse a través de la función V(...)$.... Para este ejemplo, para acceder al atributo eat_with_2 en la red net, escribimos V(net)$eat_with_2. Finalmente, individuos con eat_with_2 igual a true serán coloreados purple; de lo contrario, si es igual a FALSE, serán coloreados darkgreen. Antes de graficar las redes, veamos qué obtenemos cuando accedemos al atributo eat_with_2 en el primer grafo:
V(graphs[[1]])$eat_with_2
## [1] TRUE TRUE FALSE FALSE TRUE TRUE FALSE FALSE TRUE TRUE FALSE FALSEUn vector lógico. Ahora redibujemos las figuras:
grid.arrange(
nplot(graphs[[1]], vertex.color = color_it(graphs[[1]])),
nplot(graphs[[2]], vertex.color = color_it(graphs[[2]])),
nplot(graphs[[3]], vertex.color = color_it(graphs[[3]])),
nplot(graphs[[4]], vertex.color = color_it(graphs[[4]])),
ncol = 2, nrow = 2
)
Dado que la mayoría del tiempo, estaremos tratando con muchas redes egocéntricas; puedes querer dibujar cada red independientemente; el siguiente bloque de código hace eso. Primero, si es necesario, creará una carpeta para almacenar las redes. Luego, usando la función lapply, usará netplot::nplot() para dibujar las redes, agregar una leyenda, y guardar el grafo como .../graphml_[número].png, donde [número] irá de 01 al número total de redes en graphs.
if (!dir.exists("egonets/figs/egonets"))
dir.create("egonets/figs/egonets", recursive = TRUE)
lapply(seq_along(graphs), function(i) {
# Creando el dispositivo
png(sprintf("egonets/figs/egonets/graphml_%02i.png", i))
# Dibujando el gráfico
p <- nplot(
graphs[[i]],
vertex.color = color_it(graphs[[i]])
)
# Agregando una leyenda
p <- nplot_legend(
p,
labels = c("come con: FALSE", "come con: TRUE"),
pch = 21,
packgrob.args = list(side = "bottom"),
gp = gpar(
fill = c("darkgreen", "purple")
),
ncol = 2
)
print(p)
# Cerrando el dispositivo
dev.off()
})Archivos de persona
Como antes, listamos los archivos que terminan en Person.csv (con la ruta completa,) y los leemos en R. Aunque R tiene la función read.csv, aquí uso la función fread del paquete de R data.table. Junto con dplyr, data.table es una de las herramientas de manipulación de datos más populares en R. Además de la sintaxis, la mayor diferencia entre las dos es el rendimiento; data.table es significativamente más rápido que cualquier otro paquete de manejo de datos en R, y es una gran alternativa para manejar grandes conjuntos de datos. El siguiente bloque de código carga el paquete, lista los archivos, y los lee en R.
# Cargando data.table
library(data.table)
# Listando los archivos
person_files <- list.files(
path = "data-raw/egonets",
pattern = "*Person.csv",
full.names = TRUE
)
# Cargando todos en una sola lista
persons <- lapply(person_files, fread)
# Mirando el primer elemento
persons[[1]]
## nodeID age
## <int> <int>
## 1: 1 45
## 2: 2 32
## 3: 3 31
## 4: 4 45
## 5: 5 43
## 6: 6 47
## 7: 7 45
## 8: 8 62
## 9: 9 28
## 10: 10 41
## 11: 11 41
## 12: 12 46
## 13: 13 46
## 14: 14 46
## 15: 15 62
## 16: 16 41Una tarea común es agregar un identificador a cada conjunto de datos en persons para que sepamos a qué ego pertenecen. De nuevo, la función lapply es nuestra amiga:
persons <- lapply(seq_along(persons), function(i) {
persons[[i]][, dataset_num := i]
})En data.table, las variables se crean usando el símbolo :=. El fragmento de código anterior es equivalente a esto:
for (i in 1:length(persons)) {
persons[[i]]$dataset_num <- i
}Si es necesario, podemos transformar la lista persons en un objeto data.table (es decir, un solo data.frame) usando la función rbindlist2. El siguiente bloque de código usa esa función para combinar los data.tables en un solo conjunto de datos.
# Combinando los conjuntos de datos
persons <- rbindlist(persons)
persons
## nodeID age dataset_num
## <int> <int> <int>
## 1: 1 45 1
## 2: 2 32 1
## 3: 3 31 1
## 4: 4 45 1
## 5: 5 43 1
## ---
## 271: 7 43 19
## 272: 8 48 19
## 273: 9 70 19
## 274: 10 46 19
## 275: 11 50 19Ahora que tenemos un solo conjunto de datos, podemos hacer algo de exploración de datos. Por ejemplo, podemos usar el paquete ggplot2 para dibujar un histograma de las edades de los alters.
# Cargando el paquete ggplot2
library(ggplot2)
# Histograma de edad
ggplot(persons, aes(x = age)) + # Iniciando el gráfico
geom_histogram(fill = "purple") + # Agregando un histograma
labs(x = "Edad", y = "Frecuencia") + # Cambiando las etiquetas del eje x/y
labs(title = "Distribución de Edad de Alters") # Agregando un título
## `stat_bin()` using `bins = 30`. Pick better value `binwidth`.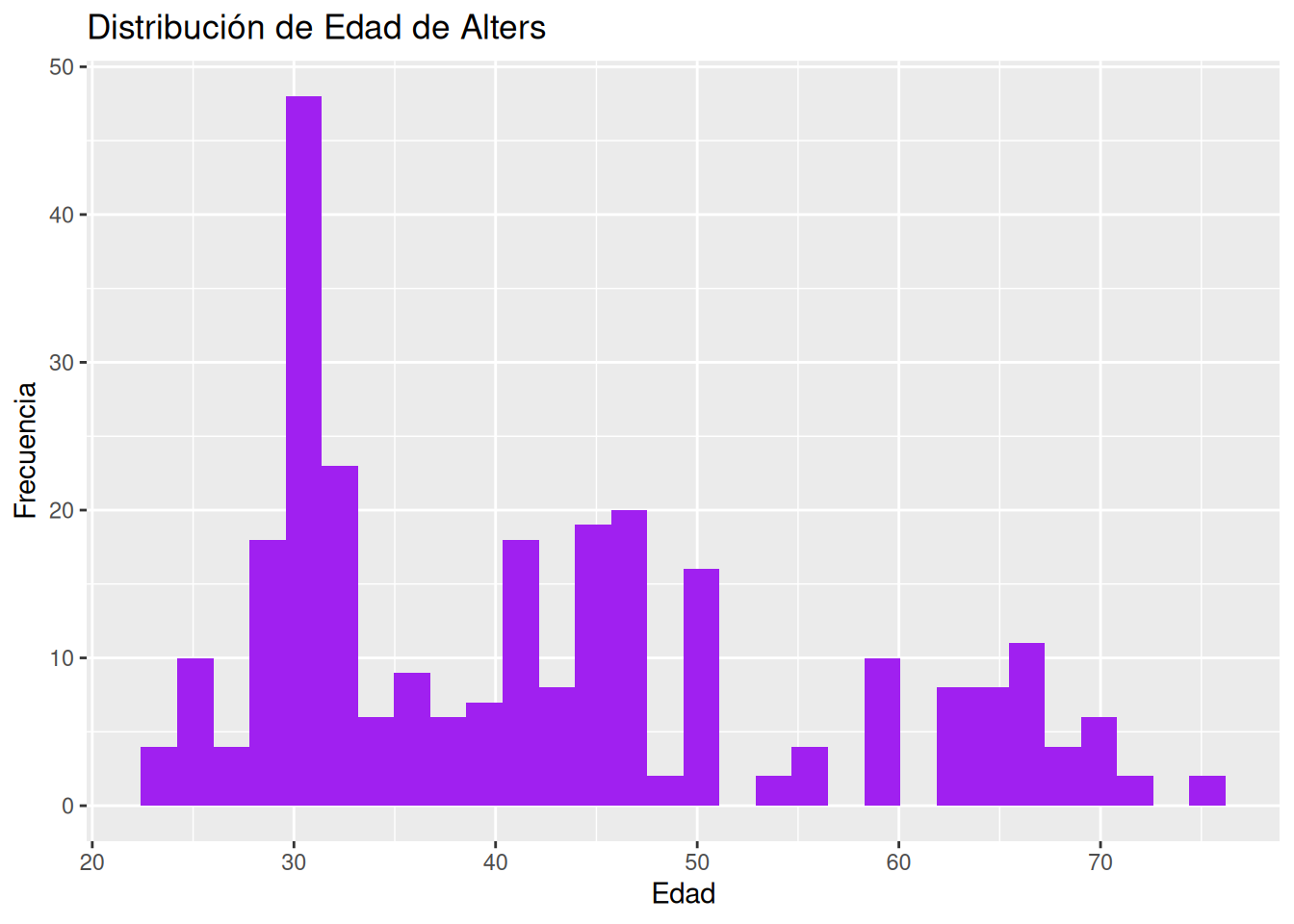
Archivos de ego
Los archivos de ego contienen información sobre los egos (¡obvio!.) De nuevo, los leeremos todos a la vez usando list.files + lapply:
# Listando archivos que terminan con *ego.csv
ego_files <- list.files(
path = "data-raw/egonets",
pattern = "*ego.csv",
full.names = TRUE
)
# Leyendo los archivos con fread
egos <- lapply(ego_files, fread)
# Combinándolos
egos <- rbindlist(egos)
head(egos)
## networkCanvasEgoUUID networkCanvasCaseID
## <char> <char>
## 1: I-11ca3a78c-62f131f37169-c139217a1f6 I_-59190_BRB9111
## 2: I-fef-ab-4-5a--7-35c4f23-96eb32-34ea I-100BB_00B95-90
## 3: I2f1bd0b6d-f71f4664cf-d-26-97408f22d I-1BB79950-0-7
## 4: Id36bb-3b2bcbd2a6239b1103134c6b3d1d6 I000091I_RB010B5
## 5: I436d32fc67fb5c6-23-244f353849b120cd I019051R0_RRR0-0
## 6: Ibf1f-2-34162bb5f2c36b8241--316a-fff I01B11-I1101_44R
## networkCanvasSessionID
## <char>
## 1: I612b7a1af---0880b-70698204-b-8dbf09
## 2: If5e0-f-26cbec070760f-e6b6d26ebfb06f
## 3: I825c293a1304-e5-cbea8a80aae05b305fa
## 4: I1b8a7d0f6b4-8298c9-848-9186d68a7f3c
## 5: Ie620be37b75983c49ac63-38-425227c959
## 6: Ie3-134323ed40-0e-d954b3d-febbcb9363
## networkCanvasProtocolName sessionStart
## <char> <POSc>
## 1: Postpartum social networks with sociogram_V5 2023-02-22 23:41:59
## 2: Postpartum social networks with sociogram_V5 2023-02-10 21:46:02
## 3: Postpartum social networks with sociogram_V5 2023-03-01 16:52:09
## 4: Postpartum social networks with sociogram_V5 2023-01-26 20:38:07
## 5: Postpartum social networks with sociogram_V5 2023-02-06 14:55:57
## 6: Postpartum social networks with sociogram_V5 2023-03-16 18:20:02
## sessionFinish sessionExported
## <POSc> <POSc>
## 1: 2023-02-23 01:47:00 2023-02-23 01:47:08
## 2: 2023-02-11 01:29:32 2023-02-11 01:34:12
## 3: 2023-03-02 16:51:20 2023-03-02 17:04:42
## 4: 2023-01-26 22:03:20 2023-01-26 22:03:34
## 5: 2023-02-06 15:49:38 2023-02-06 15:56:42
## 6: 2023-03-17 21:11:09 2023-03-17 21:16:15Algo genial sobre data.table es que, dentro de corchetes cuadrados, podemos manipular los datos refiriéndonos a las variables directamente. Por ejemplo, si quisiéramos calcular la diferencia entre sessionFinish y sessionStart, usando R base haríamos lo siguiente:
egos$total_time <- egos$sessionFinish - egos$sessionStartMientras que con data.table, la creación de variables es mucho más directa (nota que en lugar de usar <- o = para asignar una variable, usamos el operador :=):
# ¿Cuánto tiempo?
egos[, total_time := sessionFinish - sessionStart]También podemos visualizar esto usando ggplot2:
ggplot(egos, aes(x = total_time)) +
geom_histogram() +
labs(x = "Tiempo en minutos", y = "Conteo") +
labs(title = "Tiempo total gastado por egos")
## Don't know how to automatically pick scale for object of type <difftime>.
## Defaulting to continuous.
## `stat_bin()` using `bins = 30`. Pick better value `binwidth`.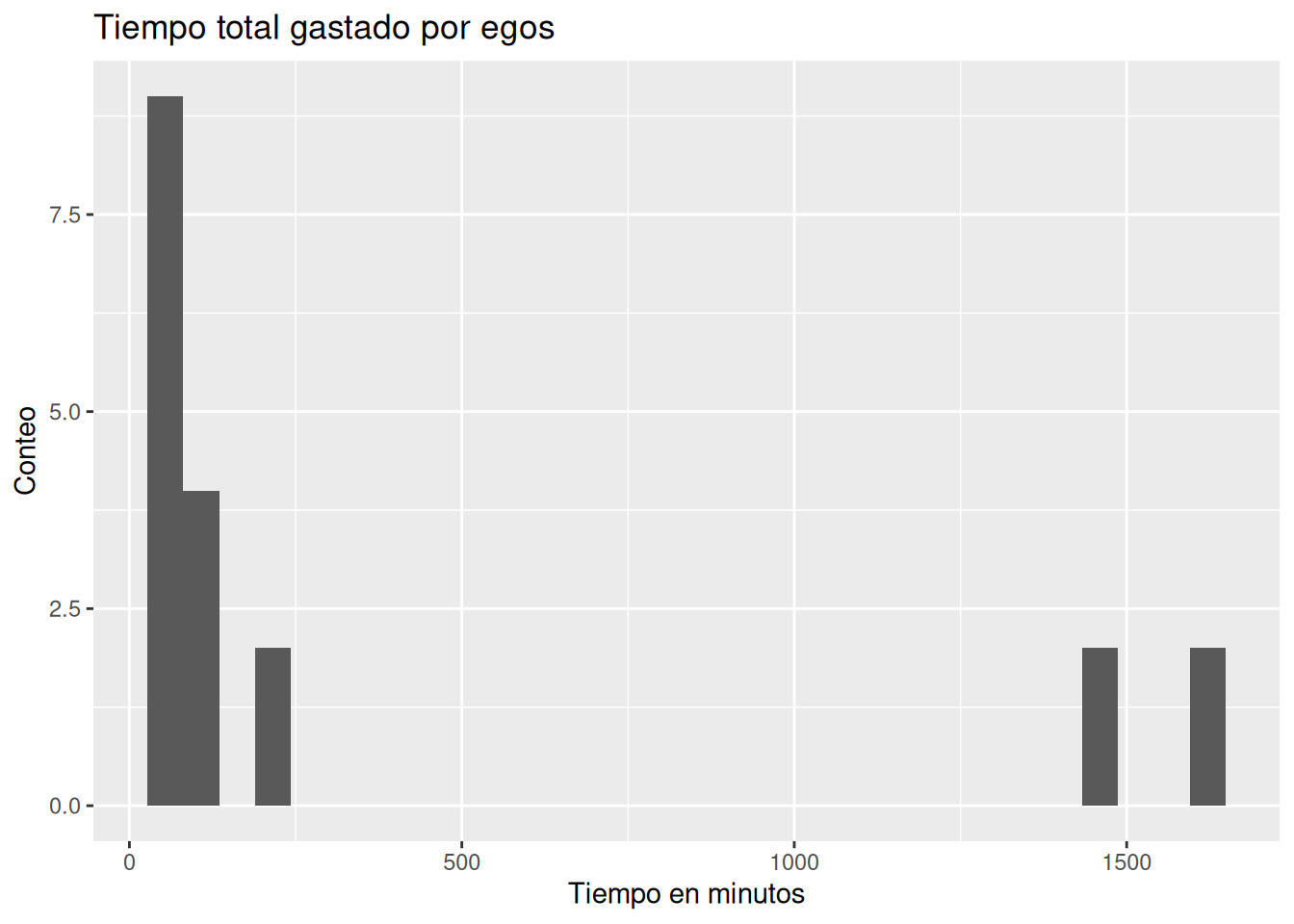
Archivos de lista de enlaces
Como mencioné antes, dado que estamos leyendo los archivos graphml, usar la lista de enlaces puede no ser necesario. Sin embargo, el proceso para importar el archivo de lista de enlaces a R es el mismo que hemos estado aplicando: listar los archivos y leerlos todos a la vez usando lapply:
# Listando todos los archivos que terminan en Knows.csv
edgelist_files <- list.files(
path = "data-raw/egonets",
pattern = "*Knows.csv",
full.names = TRUE
)
# Leyendo todos los archivos a la vez
edgelists <- lapply(edgelist_files, fread)Para evitar confusión, también podemos agregar ids correspondientes al número de archivo. Una vez que hagamos eso, podemos combinar todos los archivos en un solo objeto data.table usando rbindlist:
edgelists <- lapply(seq_along(edgelists), function(i) {
edgelists[[i]][, dataset_num := i]
})
edgelists <- rbindlist(edgelists)
head(edgelists)
## edgeID from to networkCanvasEgoUUID
## <int> <int> <int> <char>
## 1: 1 1 5 I839f-8fa8f8aeb8-eaf---ba8-cf3908f3a
## 2: 2 1 10 If81a9c0f-9f4f28ccf-c4c923a-8-0f5fce
## 3: 3 1 9 I899ffe-27-3-a3-ca2fb7f7-ca8e7715ce9
## 4: 4 1 10 I814efaba88cbb02caa8c89790-83beeaf9-
## 5: 5 7 6 Ifd-0eec2e08974eaf2b79f-9efb7e3-8998
## 6: 6 2 6 I-28fe89cc-fc5db3825b92-ae87c-c18e3d
## networkCanvasUUID networkCanvasSourceUUID
## <char> <char>
## 1: I720400eb19bccce-77cee773289b02-fe7e I4d5--16a08f8ba463c6458f8979e-65fa9d
## 2: I-b469c0-60f8bbb543-32-628-216f9-038 I-6cf8-f3da-4-96-87efaf5daaa48ba5e5c
## 3: Ifa4933-9baaf5fc-f-e4f5c5e5-ff34-f-f I5-f69a6eaa-5956e8897ca999-ffb6ed-e1
## 4: I4cb-904496b1-6194bcb51b58444b40-ef8 I3e5-6c8d5e0f086--e-5ab45-4-5aaa5-0e
## 5: I0ab7--b7a0ee71e54c1e93cdb-4ca5ab1-b I5-b-9-7eca5ab5-91915ba9b6565a6e42cc
## 6: Ic80142fc4c431009e84b3-ab3f-9b0eab03 Ie0a24eea4e01a4340343a0-66723-a-9970
## networkCanvasTargetUUID dataset_num
## <char> <int>
## 1: Id1c8befd46bdd195c-ce91a8-bc0---4f0e 1
## 2: I757b4a-3ea4d95--b9ebb9db3d55dcbaf-c 1
## 3: I92a62925ff9-e2f27-6ef97d-29fb729624 1
## 4: I7f--da48-46a64-b972c-ef6bbec--64cb4 1
## 5: I-eaa7e95659-9cf01a4f5fd69af54e6-d60 1
## 6: I69060e8a-454609-faa04cd3eeb-5-9550- 1Juntando todo
En esta última parte del capítulo, usaremos los paquetes igraph y ergm para generar características (covariables, controles, variables independientes, o como las llames) a nivel de red egocéntrica. Una vez más, la función lapply es nuestra amiga
Generando estadísticas usando igraph
El paquete de R igraph tiene múltiples rutinas de alto rendimiento para calcular estadísticas a nivel de grafo. Por ahora, nos enfocaremos en las siguientes estadísticas: conteo de vértices, conteo de enlaces, número de aislados, transitividad, y modularidad basada en centralidad de intermediación:
net_stats <- lapply(graphs, function(g) {
# Calculando modularidad
groups <- cluster_edge_betweenness(g)
# Calculando las estadísticas
data.table(
size = vcount(g),
edges = ecount(g),
nisolates = sum(degree(g) == 0),
transit = transitivity(g, type = "global"),
modular = modularity(groups)
)
})Observa que contamos aislados usando la función degree(). Podemos combinar las estadísticas en un solo data.table usando la función rbindlist:
net_stats <- rbindlist(net_stats)
head(net_stats)
## size edges nisolates transit modular
## <num> <num> <int> <num> <num>
## 1: 12 25 1 0.6750000 0.012000000
## 2: 16 47 0 0.4332130 0.003395201
## 3: 16 58 0 0.5612009 0.002675386
## 4: 15 75 0 0.8515112 0.000000000
## 5: 15 52 0 0.5780488 0.000000000
## 6: 17 68 0 0.6291161 0.025735294Generando estadísticas basadas en ergm
El paquete de R ergm tiene un conjunto mucho más grande de estadísticas a nivel de grafo que podemos agregar a nuestros modelos.3 La clave para generar estadísticas basadas en el paquete ergm es la función summary_formula. Antes de empezar a usar esa función, primero necesitamos convertir las redes igraph a objetos network, que son la clase de objeto nativa para el paquete ergm. Usamos el paquete de R intergraph para eso, y en particular, la función asNetwork:
# Cargando los paquetes requeridos
library(intergraph)
library(ergm)
## Loading required package: network
##
## 'network' 1.20.0 (2026-02-06), part of the Statnet Project
## * 'news(package="network")' for changes since last version
## * 'citation("network")' for citation information
## * 'https://statnet.org' for help, support, and other information
##
## Attaching package: 'network'
## The following objects are masked from 'package:igraph':
##
## %c%, %s%, add.edges, add.vertices, delete.edges, delete.vertices,
## get.edge.attribute, get.edges, get.vertex.attribute, is.bipartite,
## is.directed, list.edge.attributes, list.vertex.attributes,
## set.edge.attribute, set.vertex.attribute
##
## 'ergm' 4.11.0 (2025-12-22), part of the Statnet Project
## * 'news(package="ergm")' for changes since last version
## * 'citation("ergm")' for citation information
## * 'https://statnet.org' for help, support, and other information
## 'ergm' 4 is a major update that introduces some backwards-incompatible
## changes. Please type 'news(package="ergm")' for a list of major
## changes.
# Convirtiendo todos los objetos "igraph" en graphs a objetos "network"
graphs_network <- lapply(graphs, asNetwork)Con los objetos de red listos, podemos proceder a calcular estadísticas a nivel de grafo usando la función summary_formula. Aquí solo veremos: el número de triángulos, homofilia de género, y homofilia de dieta saludable:
net_stats_ergm <- lapply(graphs_network, function(n) {
# Calculando las estadísticas
s <- summary_formula(
n ~ triangles +
nodematch("gender_1") +
nodematch("healthy_diet")
)
# Guardándolas como un objeto data.table
data.table(
triangles = s[1],
gender_homoph = s[2],
healthyd_homoph = s[3]
)
})Una vez más, usamos rbindlist para combinar todas las estadísticas de red en un solo objeto data.table:
net_stats_ergm <- rbindlist(net_stats_ergm)
head(net_stats_ergm)
## triangles gender_homoph healthyd_homoph
## <num> <num> <num>
## 1: 27 11 3
## 2: 40 30 20
## 3: 81 40 29
## 4: 216 33 38
## 5: 79 44 19
## 6: 121 38 16Guardando los datos
Terminamos el capítulo guardando todo nuestro trabajo en cuatro conjuntos de datos:
Estadísticas de red (como un archivo csv)
Objetos igraph (como un archivo rda, que podemos leer de vuelta usando
read.rds)Objetos de red (ídem)
Archivos de persona (información de alters, como un archivo csv.)
Los archivos CSV pueden guardarse usando write.csv o, como hacemos aquí, fwrite del paquete data.table:
# Verificando que el directorio existe
if (!dir.exists("data"))
dir.create("data")
# Atributos de red
master <- cbind(egos, net_stats, net_stats_ergm)
fwrite(master, file = "data/network_stats.csv")
# Redes
saveRDS(graphs, file = "data/networks_igraph.rds")
saveRDS(graphs_network, file = "data/networks_network.rds")
# Atributos
fwrite(persons, file = "data/persons.csv")Agradezco a Jacqueline M. Kent-Marvick, quien me proporcionó lo que usé como línea base para generar la exportación artificial de Network Canvas.↩︎
Aunque no es lo mismo,
rbindlist(casi siempre) produce el mismo resultado que llamar la funcióndo.call. En particular, en lugar de ejecutar la llamadarbindlist(persons), podríamos haber usadodo.call(rbind, persons).↩︎¡Hay una razón obvia, los ERGMs son todo sobre estadísticas a nivel de grafo!↩︎