library(ergm)
library(netplot)
data("sampson")
nplot(samplike)
June 27, 2024
September 3, 2025
Recomiendo encarecidamente leer la viñeta del paquete de R ergm.
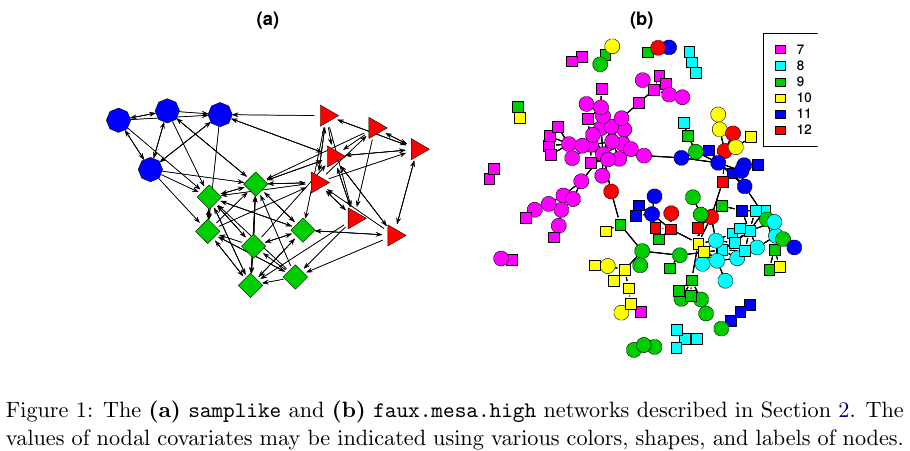
El propósito de los ERGMs, en pocas palabras, es describir de manera parsimoniosa las fuerzas de selección local que dan forma a la estructura global de una red. Para este fin, un conjunto de datos de red, como los que se muestran en la Figura 1, puede ser considerado como la respuesta en un modelo de regresión, donde los predictores son cosas como “propensión de individuos del mismo sexo a formar asociaciones” o “propensión de individuos a formar triángulos de asociaciones”. En la Figura 1(b), por ejemplo, es evidente que los nodos individuales parecen agruparse en grupos de las mismas etiquetas numéricas (que resultan ser las calificaciones de los estudiantes, del 7 al 12); por lo tanto, un ERGM puede ayudarnos a cuantificar la fuerza de este efecto intra-grupo.
En pocas palabras, usamos ERGMs (que en inglés son “Exponential Random Graph Models”) como una interpretación paramétrica de la distribución de \mathbf{Y}, que toma la forma canónica:
{\mathbb{P}_{\mathcal{Y}}\left(\mathbf{Y}=\mathbf{y};\mathbf{\theta}\right) } = \frac{\text{exp}\left\{\theta^{\text{T}}\mathbf{g}(\mathbf{y})\right\}}{\kappa\left(\theta, \mathcal{Y}\right)},\quad\mathbf{y}\in\mathcal{Y} \tag{9.1}
Donde \theta\in\Omega\subset\mathbb{R}^q es el vector de coeficientes del modelo y \mathbf{g}(\mathbf{y}) es un q-vector de estadísticas basadas en la matriz de adyacencia \mathbf{y}.
El modelo Equation 9.1 puede expandirse reemplazando \mathbf{g}(\mathbf{y}) con \mathbf{g}(\mathbf{y}, \mathbf{X}) para permitir información adicional de covariables \mathbf{X} sobre la red. El denominador \kappa\left(\theta,\mathcal{Y}\right) = \sum_{\mathbf{y}\in\mathcal{Y}}\text{exp}\left\{\theta^{\text{T}}\mathbf{g}(\mathbf{y})\right\} es el factor normalizador que asegura que la ecuación Equation 9.1 sea una distribución de probabilidad legítima. Incluso después de fijar \mathcal{Y} para que sean todas las redes que tienen tamaño n, el tamaño de \mathcal{Y} hace que este tipo de modelo estadístico sea difícil de estimar ya que hay N = 2^{n(n-1)} redes posibles! (David R. Hunter et al. 2008)
Desarrollos posteriores incluyen nuevas estructuras de dependencia para considerar efectos de vecindario más generales. Estos modelos relajan las suposiciones de dependencia markoviana de un paso, permitiendo la investigación de configuraciones de mayor alcance, como rutas más largas en la red o ciclos más grandes (Pattison y Robins 2002). Se han desarrollado modelos para estructuras de red bipartitas (Faust y Skvoretz 1999) y tripartitas (Mische y Robins 2000). (David R. Hunter et al. 2008, 9)
En el caso más simple, los ERGMs equivalen a una regresión logística. Por simple, me refiero a casos sin términos markovianos–motivos que involucran más de un enlace–por ejemplo, el grafo de Bernoulli. En el grafo de Bernoulli, los vínculos son independientes, por lo que la presencia/ausencia de un vínculo entre los nodos i y j no afectará la presencia/ausencia de un vínculo entre los nodos k y l.
Ajustemos un ERGM usando el conjunto de datos sampson en el paquete ergm.
Usar ergm para ajustar un grafo de Bernoulli requiere usar el término edges, que cuenta cuántos vínculos hay en el grafo:
Dado que esto es equivalente a una regresión logística, podemos usar la función glm para ajustar el mismo modelo. Primero, necesitamos preparar los datos para que podamos pasarlos a glm:
y <- sort(as.vector(as.matrix(samplike)))
y <- y[-c(1:18)] # Eliminamos la diagonal del modelo, que es todo 0.
y
## [1] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## [38] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## [75] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## [112] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## [149] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## [186] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 1 1
## [223] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [260] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [297] 1 1 1 1 1 1 1 1 1 1Ahora podemos ajustar el modelo GLM:
Los coeficientes de ambos ERGM y GLM deberían coincidir:
glm_fit
##
## Call: glm(formula = y ~ 1, family = binomial("logit"))
##
## Coefficients:
## (Intercept)
## -0.9072
##
## Degrees of Freedom: 305 Total (i.e. Null); 305 Residual
## Null Deviance: 367.2
## Residual Deviance: 367.2 AIC: 369.2
ergm_fit
##
## Call:
## ergm(formula = samplike ~ edges)
##
## Maximum Likelihood Coefficients:
## edges
## -0.9072Además, en el caso del grafo de Bernoulli, podemos obtener la estimación usando la función Logit:
De nuevo, el mismo resultado. El grafo de Bernoulli no es el único modelo ERGM que puede ajustarse usando una regresión logística. Además, si todos los términos del modelo son términos no-Markov, ergm automáticamente usa por defecto una regresión logística.
El objetivo final es realizar inferencia estadística sobre el modelo propuesto. En un entorno estándar, podríamos usar Estimación de Máxima Verosimilitud (MLE, por sus siglas en inglés), que consiste en encontrar los parámetros del modelo \theta que, dados los datos observados, maximicen la verosimilitud del modelo. Para esto último, generalmente usamos el método de Newton. El método de Newton requiere calcular la log-verosimilitud del modelo, lo que puede ser desafiante en ERGMs.
Para ERGMs, dado que parte de la verosimilitud involucra una constante normalizadora que es una función de todas las redes posibles, esto no es tan directo como en el entorno regular. Debido a esto, la mayoría de los métodos de estimación se basan en simulaciones.
En statnet, el método de estimación predeterminado se basa en un método propuesto por (Geyer and Thompson 1992), MLE de Cadena de Markov, que usa Monte Carlo de Cadena de Markov para simular redes y una versión modificada del algoritmo Newton-Raphson para estimar los parámetros.
La idea del MC-MLE para esta familia de modelos estadísticos es aproximar la expectativa de las razones de constantes normalizadoras usando la ley de los grandes números. En particular, lo siguiente:
\begin{align*} \frac{\kappa\left(\theta,\mathcal{Y}\right)}{\kappa\left(\theta_0,\mathcal{Y}\right)} & = \frac{ \sum_{\mathbf{y}\in\mathcal{Y}}\text{exp}\left\{\theta^{\text{T}}\mathbf{g}(\mathbf{y})\right\}}{ \sum_{\mathbf{y}\in\mathcal{Y}}\text{exp}\left\{\theta_0^{\text{T}}\mathbf{g}(\mathbf{y})\right\} } \\ & = \sum_{\mathbf{y}\in\mathcal{Y}}\left( % \frac{1}{% \sum_{\mathbf{y}\in\mathcal{Y}\text{exp}\left\{\theta_0^{\text{T}}\mathbf{g}(\mathbf{y})\right\}}% } \times % \text{exp}\left\{\theta^{\text{T}}\mathbf{g}(\mathbf{y})\right\} % \right) \\ & = \sum_{\mathbf{y}\in\mathcal{Y}}\left( % \frac{\text{exp}\left\{\theta_0^{\text{T}}\mathbf{g}(\mathbf{y})\right\}}{% \sum_{\mathbf{y}\in\mathcal{Y}\text{exp}\left\{\theta_0^{\text{T}}\mathbf{g}(\mathbf{y})\right\}}% } \times % \text{exp}\left\{(\theta - \theta_0)^{\text{T}}\mathbf{g}(\mathbf{y})\right\} % \right) \\ & = \sum_{\mathbf{y}\in\mathcal{Y}}\left( % {\mathbb{P}_{\times }\left(Y = y|\mathcal{Y}, \theta_0\right) }% \text{exp}\left\{(\theta - \theta_0)^{\text{T}}\mathbf{g}(\mathbf{y})\right\} % \right) \\ & = \text{E}_{\theta_0}\left(\text{exp}\left\{(\theta - \theta_0)^{\text{T}}\mathbf{g}(\mathbf{y})\right\} \right) \end{align*}
En particular, el algoritmo MC-MLE usa este hecho para maximizar la razón de log-verosimilitud. La función objetivo misma puede aproximarse simulando m redes de la distribución con parámetro \theta_0:
l(\theta) - l(\theta_0) \approx (\theta - \theta_0)^{\text{T}}\mathbf{g}(\mathbf{y}_{obs}) - \text{log}{\left[\frac{1}{m}\sum_{i = 1}^m\text{exp}\left\{(\theta-\theta_0)^{\text{T}}\right\}\mathbf{g}(\mathbf{Y}_i)\right]}
Para más detalles, ver (David R. Hunter et al. 2008). Un bosquejo del algoritmo sigue:
Inicializar el algoritmo con una conjetura inicial de \theta, llamarlo \theta^{(t)} (debe ser una conjetura bastante buena)
Mientras (no haya convergencia) hacer:
Usando \theta^{(t)}, simular M redes por medio de pequeños cambios en la \mathbf{Y}_{obs} (la red observada). Esta parte se hace usando un método de muestreo de importancia que pondera cada red propuesta por su verosimilitud condicional en \theta^{(t)}
Con las redes simuladas, podemos hacer el paso de Newton para actualizar el parámetro \theta^{(t)} (esta es la parte de iteración en el paquete ergm): \theta^{(t)}\to\theta^{(t+1)}.
Si se ha alcanzado la convergencia (lo que usualmente significa que \theta^{(t)} y \theta^{(t + 1)} no son muy diferentes), entonces parar; de lo contrario, ir al paso a.
Lusher, Koskinen, and Robins (2013);Admiraal and Handcock (2006);Snijders (2002);Wang et al. (2009) proporcionan detalles sobre el algoritmo usado por PNet (el mismo que el usado en RSiena), y Lusher, Koskinen, and Robins (2013) proporciona una breve discusión sobre las diferencias entre ergm y PNet.
ergmDe la sección anterior:1
En esta sección, usaremos el paquete ergm (del conjunto de paquetes statnet Handcock et al. (2023),) y el paquete intergraph (Bojanowski 2023). Este último proporciona funciones para ir y venir entre objetos igraph y network de los paquetes igraph y network respectivamente2
Como una nota lateral bastante importante, el orden en que se cargan los paquetes de R importa. ¿Por qué es importante mencionarlo ahora? Bueno, resulta que al menos un par de funciones en el paquete network tienen el mismo nombre que algunas funciones en el paquete igraph. Cuando se carga el paquete ergm, dado que depende de network, cargará el paquete network primero, lo que enmascarará algunas funciones en igraph. Esto se vuelve evidente una vez que cargas ergm después de cargar igraph:
Los siguientes objetos están enmascarados desde 'package:igraph':
add.edges, add.vertices, %c%, delete.edges, delete.vertices, get.edge.attribute, get.edges,
get.vertex.attribute, is.bipartite, is.directed, list.edge.attributes, list.vertex.attributes, %s%,
set.edge.attribute, set.vertex.attribute¿Cuáles son las implicaciones de esto? Si llamas la función list.edge.attributes para un objeto de clase igraph R devolverá un error ya que la primera función que coincide con ese nombre viene del paquete network! Para evitar esto puedes usar la notación de doble dos puntos:
Usando la función asNetwork, podemos coercionar el objeto igraph en un objeto network para que podamos usarlo con la función ergm:
# Creando la nueva red
network_111 <- intergraph::asNetwork(ig_year1_111)
# Ejecutando un ergm simple (solo ajustando cuenta de enlaces)
ergm(network_111 ~ edges)
## Warning in ergm.getnetwork(formula): This network contains loops
## Starting maximum pseudolikelihood estimation (MPLE):
## Obtaining the responsible dyads.
## Evaluating the predictor and response matrix.
## Maximizing the pseudolikelihood.
## Finished MPLE.
## Evaluating log-likelihood at the estimate.
##
## Call:
## ergm(formula = network_111 ~ edges)
##
## Maximum Likelihood Coefficients:
## edges
## -4.734Entonces, ¿qué pasó aquí? Obtuvimos una advertencia. Resulta que nuestra red tiene bucles (¡no pensé en eso antes!), que son arcos que conectan un nodo consigo mismo. Echemos un vistazo a eso con la función which_loop
Podemos deshacernos de estos usando el igraph::-.igraph. Eliminemos los aislados usando el mismo operador
# Creando la nueva red
network_111 <- ig_year1_111
# Eliminando bucles
network_111 <- network_111 - E(network_111)[which(which_loop(network_111))]
# Eliminando aislados
network_111 <- network_111 - which(degree(network_111, mode = "all") == 0)
# Convirtiendo la red
network_111 <- intergraph::asNetwork(network_111)asNetwork(simplify(ig_year1_111)) ig_year1_111 |> simplify() |> asNetwork()
Un problema que tenemos con estos datos es el hecho de que algunos vértices tienen valores faltantes en las variables hispanic, female1, y eversmk1. Por ahora, procederemos imputando valores basados en los promedios:
Echemos un vistazo a la red
Colorando por hispanic, podemos ver que los nodos parecen estar agrupados por este atributo. La inspección visual en la ciencia de redes puede ser muy útil, pero, para hacer declaraciones más formales, necesitamos usar modelos estadísticos; aquí es donde los ERGMs brillan.
Aunque no existe una solución única para estimar estos modelos, el siguiente flujo de trabajo me ha funcionado en el pasado:
Estimar el modelo más simple, agregando una variable a la vez.
Después de cada estimación, ejecutar la función mcmc.diagnostics para ver qué tan bien (o mal) se comportaron las cadenas.
Ejecutar la función gof y verificar qué tan bien el modelo coincide con las estadísticas estructurales de la red.
Qué usar:
control.ergms: Número máximo de iteraciones, semilla para Pseudo-RNG, cuántos núcleos
ergm.constraints: De dónde muestrear la red. Da estabilidad y (en algunos casos) convergencia más rápida ya que al restringir el modelo estás reduciendo el tamaño de la muestra.
Aquí hay un ejemplo de un par de modelos que podríamos comparar
Nota que este documento puede no incluir los mensajes usuales que el comando ergm genera durante el procedimiento de estimación. Esto es solo para hacerlo más amigable para imprimir.
ans0 <- ergm(
network_111 ~
edges +
nodematch("hispanic") +
nodematch("female1") +
nodematch("eversmk1") +
mutual,
control = control.ergm(
seed = 1,
MCMLE.maxit = 10,
parallel = 4,
CD.maxit = 10
)
)
## Warning: 'glpk' selected as the solver, but package 'Rglpk' is not available;
## falling back to 'lpSolveAPI'. This should be fine unless the sample size and/or
## the number of parameters is very big.En una versión anterior del libro usamos la restricción bd para limitar el número máximo de vínculos salientes a 19. Aunque esta es una restricción razonable (ya que la muestra pudo haber sido restringida por construcción), la versión actual del paquete ERGM devuelve una advertencia y muestra las estadísticas GOF con signos cambiados (AIC y BIC negativos, y Logverosimilitud positiva).
Entonces, ¿qué estamos haciendo aquí:
El modelo está controlando por:
edges Número de enlaces en la red (en oposición a su densidad)
nodematch("algún-nombre-de-variable-aquí") Incluye un término que controla por homofilia/heterofilia
mutual Número de conexiones mutuas entre (i, j), (j, i). Esto puede estar relacionado con, por ejemplo, cierre triádico.
Para más sobre parámetros de control, ver Morris, Handcock, and Hunter (2008). El siguiente modelo consiste en una version más simple del modelo anterior. Excluye el término mutual lleva a un modelo sin términos markovianos, es decir, los estadísticos de la red son funciones de enlaces individuales y no de pares de enlaces o más. El término mutual incluye dos enlaces (i\to j y j\to i). Modelos sin términos markovianos pueden ser estimados usando regresión logística, evitando la necesidad de simulaciones MCMC:
Finalmente, podemos intentar estimar un modelo con términos más complejos, por ejemplo, el geometrically weighted edge-wise shared partner (gwesp). Éste término captura el cierre triádico, pero es más fácil de estimar que triangle. Más aún, para facilitar el proceso de estimación, podemos incorporar el termino geometrically weighted dyad-wise shared partner (gwdsp). Ambos estadísticos incluyen un parámetro de descuento (decay); por el momento, fijaremos ambos parametros en 0.5 (valores entre 0.25 y 1.5 son comunes). Más detalles sobre estos términos en David R. Hunter (2007).
Ahora, un truco agradable para ver todas las regresiones en la misma tabla, podemos usar el paquete texreg (Leifeld 2013) que soporta salidas de ergm!
library(texreg)
## Version: 1.39.5
## Date: 2025-12-21
## Author: Philip Leifeld (University of Manchester)
##
## Consider submitting praise using the praise or praise_interactive functions.
## Please cite the JSS article in your publications -- see citation("texreg").
screenreg(list(ans0, ans1, ans2))
##
## ================================================================
## Model 1 Model 2 Model 3
## ----------------------------------------------------------------
## edges -5.62 *** -5.49 *** -4.73 ***
## (0.05) (0.06) (0.07)
## nodematch.hispanic 0.22 *** 0.30 *** 0.16 ***
## (0.04) (0.05) (0.03)
## nodematch.female1 0.87 *** 1.16 *** 0.47 ***
## (0.04) (0.05) (0.03)
## nodematch.eversmk1 0.33 *** 0.45 *** 0.23 ***
## (0.04) (0.04) (0.03)
## mutual 4.10 *** 2.72 ***
## (0.07) (0.08)
## gwesp.OTP.fixed.0.5 1.34 ***
## (0.03)
## gwdsp.OTP.fixed.0.5 -0.11 ***
## (0.01)
## ----------------------------------------------------------------
## AIC 22649.55 25135.63 20155.83
## BIC 22699.89 25175.91 20226.31
## Log Likelihood -11319.77 -12563.82 -10070.91
## ================================================================
## *** p < 0.001; ** p < 0.01; * p < 0.05O, si estás usando rmarkdown, puedes exportar los resultados usando LaTeX o html, intentemos este último para ver cómo se ve aquí:
| Model 1 | Model 2 | Model 3 | |
|---|---|---|---|
| edges | -5.62*** | -5.49*** | -4.73*** |
| (0.05) | (0.06) | (0.07) | |
| nodematch.hispanic | 0.22*** | 0.30*** | 0.16*** |
| (0.04) | (0.05) | (0.03) | |
| nodematch.female1 | 0.87*** | 1.16*** | 0.47*** |
| (0.04) | (0.05) | (0.03) | |
| nodematch.eversmk1 | 0.33*** | 0.45*** | 0.23*** |
| (0.04) | (0.04) | (0.03) | |
| mutual | 4.10*** | 2.72*** | |
| (0.07) | (0.08) | ||
| gwesp.OTP.fixed.0.5 | 1.34*** | ||
| (0.03) | |||
| gwdsp.OTP.fixed.0.5 | -0.11*** | ||
| (0.01) | |||
| AIC | 22649.55 | 25135.63 | 20155.83 |
| BIC | 22699.89 | 25175.91 | 20226.31 |
| Log Likelihood | -11319.77 | -12563.82 | -10070.91 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |||
En base a los resultados, podemos hacer las siguientes observaciones:
De los tres modelos, el último es el mejor en términos de menor AIC y BIC, y mayor Logverosimilitud.
Todos los términos nodematch son significativos y positivos, lo que significa que tenemos homofilia por hispanic, female1, y eversmk1.
El término mutual es significativo, positivo y grande (~4). Esto indica una fuerte tendencia de los individuos a establecer amistad mutua.
El término gwesp es positivo y significativo. La interpretación de este término no siempre es simple. Generalmente, decimos que un término gwesp positivo significa que hay una tendencia hacia el cierre triádico.
En términos brutos, una vez que cada cadena ha alcanzado la distribución estacionaria, podemos decir que no hay problemas con la autocorrelación y que cada punto de muestra es iid. Esto último implica que, dado que estamos ejecutando el modelo con más de una cadena, podemos usar todas las muestras (cadenas) como un solo conjunto de datos.
Cambios recientes en el algoritmo de estimación de ergm significan que estos gráficos ya no pueden usarse para asegurar que las estadísticas medias del modelo coincidan con las estadísticas de red observadas. Para esa funcionalidad, por favor usa el comando GOF:
gof(object, GOF=~model).—?ergm::mcmc.diagnostics
Dado que ans2 es el mejor modelo, veamos las estadísticas GOF. Primero, veamos cómo se comportó el MCMC. Podemos usar la función mcmc.diagnostics incluida en el paquete. La función es un envoltorio de un par de funciones del paquete coda (Plummer et al. 2006), que son llamadas sobre el objeto $sample que contiene las estadísticas centradas de las redes muestreadas. Al principio, puede ser confuso mirar el objeto $sample; no coincide ni con las estadísticas observadas ni con los coeficientes.
Cuando se llama mcmc.diagnostics(ans2, centered = FALSE), verás muchas salidas, incluyendo un par de gráficos mostrando la traza y distribución posterior de las estadísticas no centradas (centered = FALSE). Los siguientes fragmentos de código reproducirán la salida de la función mcmc.diagnostics paso a paso usando el paquete coda. Primero, necesitamos descentrar el objeto de muestra:
# Obteniendo la muestra centrada
sample_centered <- ans2$sample
# Obteniendo las estadísticas observadas y convirtiéndolas en una matriz para que podamos agregarlas
# a las muestras
observed <- summary(ans2$formula)
observed <- matrix(
observed,
nrow = nrow(sample_centered[[1]]),
ncol = length(observed),
byrow = TRUE
)
# Ahora descentramos la muestra
sample_uncentered <- lapply(sample_centered, function(x) {
x + observed
})
# Tenemos que hacer de esto un objeto mcmc.list
sample_uncentered <- coda::mcmc.list(sample_uncentered)Bajo el capó:
summary(sample_uncentered)
##
## Iterations = 327680:5963776
## Thinning interval = 32768
## Number of chains = 4
## Sample size per chain = 173
##
## 1. Empirical mean and standard deviation for each variable,
## plus standard error of the mean:
##
## Mean SD Naive SE Time-series SE
## edges 2471.9 63.94 2.430 6.221
## nodematch.hispanic 1829.8 59.02 2.244 7.099
## nodematch.female1 1874.4 71.09 2.703 8.346
## nodematch.eversmk1 1755.6 58.02 2.206 6.746
## mutual 481.9 27.72 1.054 3.379
## gwesp.OTP.fixed.0.5 1762.6 113.98 4.333 13.574
## gwdsp.OTP.fixed.0.5 13175.1 644.91 24.516 66.387
##
## 2. Quantiles for each variable:
##
## 2.5% 25% 50% 75% 97.5%
## edges 2347.3 2431 2469 2510 2605
## nodematch.hispanic 1718.3 1791 1827 1867 1955
## nodematch.female1 1736.0 1827 1871 1928 2016
## nodematch.eversmk1 1654.3 1714 1752 1794 1879
## mutual 429.3 462 481 500 542
## gwesp.OTP.fixed.0.5 1540.9 1684 1760 1835 2013
## gwdsp.OTP.fixed.0.5 11958.3 12737 13137 13596 14488coda::crosscorr(sample_uncentered)
## edges nodematch.hispanic nodematch.female1
## edges 1.0000000 0.8566830 0.8770210
## nodematch.hispanic 0.8566830 1.0000000 0.7573169
## nodematch.female1 0.8770210 0.7573169 1.0000000
## nodematch.eversmk1 0.8229030 0.6949758 0.7455362
## mutual 0.7921269 0.7019044 0.7765035
## gwesp.OTP.fixed.0.5 0.8782943 0.7780147 0.8679069
## gwdsp.OTP.fixed.0.5 0.9678268 0.8370254 0.8591945
## nodematch.eversmk1 mutual gwesp.OTP.fixed.0.5
## edges 0.8229030 0.7921269 0.8782943
## nodematch.hispanic 0.6949758 0.7019044 0.7780147
## nodematch.female1 0.7455362 0.7765035 0.8679069
## nodematch.eversmk1 1.0000000 0.7067807 0.7820246
## mutual 0.7067807 1.0000000 0.8954091
## gwesp.OTP.fixed.0.5 0.7820246 0.8954091 1.0000000
## gwdsp.OTP.fixed.0.5 0.8098323 0.7688635 0.8575789
## gwdsp.OTP.fixed.0.5
## edges 0.9678268
## nodematch.hispanic 0.8370254
## nodematch.female1 0.8591945
## nodematch.eversmk1 0.8098323
## mutual 0.7688635
## gwesp.OTP.fixed.0.5 0.8575789
## gwdsp.OTP.fixed.0.5 1.0000000coda::autocorr(sample_uncentered)[[1]]
## , , edges
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 1.000000000 0.85174677 0.90035143 0.84731435
## Lag 32768 0.604509897 0.52958876 0.66467595 0.54146395
## Lag 163840 0.369212891 0.35412936 0.40011462 0.39283115
## Lag 327680 0.184463035 0.14706318 0.24305479 0.29114538
## Lag 1638400 0.009007472 -0.02631294 0.03456016 -0.04578478
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 0.83296982 0.9136663 0.97089215
## Lag 32768 0.68928494 0.6870541 0.57938173
## Lag 163840 0.45203629 0.4511364 0.36536136
## Lag 327680 0.12747154 0.1829741 0.15036241
## Lag 1638400 0.05607638 0.0292786 -0.01022981
##
## , , nodematch.hispanic
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 0.851746774 1.00000000 0.796948280 0.77244980
## Lag 32768 0.540210932 0.64168452 0.620791472 0.51899205
## Lag 163840 0.348421398 0.40142712 0.409478507 0.40705581
## Lag 327680 0.156894106 0.10147048 0.226595532 0.24435622
## Lag 1638400 0.007637041 -0.04655592 -0.007443898 -0.07226516
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 0.69025542 0.795564260 0.82655175
## Lag 32768 0.58418987 0.623959752 0.52242442
## Lag 163840 0.42435250 0.432709863 0.35047668
## Lag 327680 0.11948613 0.174673754 0.13223947
## Lag 1638400 0.05123385 0.007771714 -0.02509792
##
## , , nodematch.female1
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 0.90035143 0.796948280 1.00000000 0.79308088
## Lag 32768 0.60258009 0.536790742 0.73348572 0.56527326
## Lag 163840 0.32695326 0.324827911 0.37903351 0.38465484
## Lag 327680 0.11123941 0.111356563 0.21164986 0.27852000
## Lag 1638400 0.07516871 -0.002982911 0.09513563 -0.06106261
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 0.79999770 0.88951827 0.87107087
## Lag 32768 0.66026068 0.69041074 0.56750224
## Lag 163840 0.38096094 0.39655308 0.32909183
## Lag 327680 0.07195343 0.12801382 0.08708550
## Lag 1638400 0.11308394 0.09505784 0.04044172
##
## , , nodematch.eversmk1
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 0.84731435 0.7724498 0.79308088 1.0000000
## Lag 32768 0.54365021 0.5122084 0.63731624 0.6624233
## Lag 163840 0.31661036 0.3735145 0.38613833 0.4631210
## Lag 327680 0.12487907 0.1838576 0.21306300 0.2895293
## Lag 1638400 -0.04843904 -0.0871528 -0.05860075 -0.1732522
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 0.73949417 0.82596806 0.82962588
## Lag 32768 0.59917271 0.64206417 0.53636921
## Lag 163840 0.36156912 0.38961111 0.32502461
## Lag 327680 0.06513275 0.10756201 0.12157020
## Lag 1638400 -0.02126220 -0.05784788 -0.05300152
##
## , , mutual
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 0.832969818 0.6902554 0.79999770 0.73949417
## Lag 32768 0.655957685 0.5420351 0.68082349 0.60650896
## Lag 163840 0.453711697 0.3807566 0.46832909 0.47654529
## Lag 327680 0.189592365 0.1950812 0.25444882 0.34592646
## Lag 1638400 0.004326463 -0.0397559 0.03453263 -0.04956656
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 1.00000000 0.91166903 0.790126039
## Lag 32768 0.77089766 0.75016262 0.613992396
## Lag 163840 0.51073753 0.53492576 0.445401161
## Lag 327680 0.19116258 0.22568907 0.144338541
## Lag 1638400 0.01557354 0.02072621 -0.002615637
##
## , , gwesp.OTP.fixed.0.5
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 0.91366631 0.79556426 0.88951827 0.82596806
## Lag 32768 0.65221015 0.57748172 0.71103822 0.63534710
## Lag 163840 0.40355191 0.36375124 0.42906065 0.45848417
## Lag 327680 0.16182896 0.15751100 0.23871746 0.30543634
## Lag 1638400 0.02774368 -0.01550744 0.04360474 -0.05912761
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 0.91166903 1.00000000 0.88286005
## Lag 32768 0.75714027 0.74743488 0.62020864
## Lag 163840 0.47254922 0.48618158 0.40054594
## Lag 327680 0.14530942 0.18887509 0.12356707
## Lag 1638400 0.05355936 0.04005611 0.01544559
##
## , , gwdsp.OTP.fixed.0.5
##
## edges nodematch.hispanic nodematch.female1 nodematch.eversmk1
## Lag 0 0.97089215 0.82655175 0.87107087 0.82962588
## Lag 32768 0.56904543 0.49933571 0.63747170 0.51566990
## Lag 163840 0.31817376 0.32249022 0.37414872 0.32999678
## Lag 327680 0.15987338 0.11941690 0.21123819 0.24152798
## Lag 1638400 0.01040193 -0.02676337 0.01558998 -0.05445063
## mutual gwesp.OTP.fixed.0.5 gwdsp.OTP.fixed.0.5
## Lag 0 0.79012604 0.8828601 1.00000000
## Lag 32768 0.64720629 0.6437981 0.56197239
## Lag 163840 0.40245842 0.4041699 0.32130564
## Lag 327680 0.09213271 0.1437246 0.12697167
## Lag 1638400 0.06161009 0.0289774 -0.01011366“Si las muestras se extraen de la distribución estacionaria de la cadena, las dos medias son iguales y la estadística de Geweke tiene una distribución normal estándar asintóticamente. […] El Z-score se calcula bajo la suposición de que las dos partes de la cadena son asintóticamente independientes, lo que requiere que la suma de frac1 y frac2 sea estrictamente menor que 1.”
—?coda::geweke.diag
Echemos un vistazo a una sola cadena:
Gelman y Rubin (1992) proponen un enfoque general para monitorear la convergencia de salida MCMC en el que se ejecutan m > 1 cadenas paralelas con valores iniciales que están sobre-dispersos relativo a la distribución posterior. La convergencia se diagnostica cuando las cadenas han ‘olvidado’ sus valores iniciales, y la salida de todas las cadenas es indistinguible. El diagnóstico gelman.diag se aplica a una sola variable de la cadena. Se basa en una comparación de varianzas dentro de cadena y entre cadenas, y es similar a un análisis de varianza clásico. —?coda::gelman.diag
Como diferencia del estadístico de diagnóstico anterior, este usa todas las cadenas simultáneamente:
coda::gelman.diag(sample_uncentered)
## Potential scale reduction factors:
##
## Point est. Upper C.I.
## edges 1.05 1.14
## nodematch.hispanic 1.02 1.04
## nodematch.female1 1.07 1.20
## nodematch.eversmk1 1.07 1.22
## mutual 1.07 1.20
## gwesp.OTP.fixed.0.5 1.09 1.26
## gwdsp.OTP.fixed.0.5 1.06 1.18
##
## Multivariate psrf
##
## 1.15Como regla general, valores en el [.9,1.1] son buenos.
Una característica agradable de la función mcmc.diagnostics son los gráficos bonitos de traza y distribución posterior que genera. Si tienes el paquete de R latticeExtra (Sarkar and Andrews 2022), la función anulará los gráficos predeterminados usados por coda::plot.mcmc y usará lattice en su lugar, creando gráficos de mejor apariencia. El siguiente fragmento de código llama la función mcmc.diagnostic, pero suprimimos el resto de la salida (ver figura ?fig-coda-plots).
Si llamamos la función mcmc.diagnostics, este mensaje aparece al final:
Los diagnósticos MCMC mostrados aquí son de la última ronda de simulación, previo al cálculo de las estimaciones finales de parámetros. Porque las estimaciones finales son refinamientos de aquellas usadas para esta ejecución de simulación, estos diagnósticos pueden subestimar el rendimiento del modelo. Para evaluar directamente el rendimiento del modelo final en estadísticas del modelo, por favor usa el comando GOF: gof(ergmFitObject, GOF=~model).
—
mcmc.diagnostics(ans0)
¡No está tan mal (aunque el término mutual podría hacerlo mejor)!3 Primero, observa que en la figura, vemos cuatro líneas diferentes; ¿por qué es eso? Dado que estábamos ejecutando en paralelo usando cuatro núcleos, el algoritmo ejecutó cuatro cadenas del algoritmo MCMC. Una prueba visual es ver si todas las cadenas se movieron más o menos al mismo lugar; en tal caso, podemos empezar a pensar sobre convergencia del modelo desde la perspectiva MCMC.
Una vez que estamos seguros de haber alcanzado convergencia en el algoritmo MCMC, podemos empezar a pensar sobre qué tan bien nuestro modelo predice las propiedades de la red observada. Además de las estadísticas que definen nuestro ERGM, el comportamiento predeterminado de la función gof muestra GOF para:
Echemos un vistazo
# Calculando e imprimiendo estadísticas GOF
ans_gof <- gof(ans2)
ans_gof
##
## Goodness-of-fit for model statistics
##
## obs min mean max MC p-value
## edges 2475.000 2294.000 2423.050 2622.000 0.44
## nodematch.hispanic 1832.000 1675.000 1804.840 1944.000 0.66
## nodematch.female1 1879.000 1702.000 1810.930 1933.000 0.26
## nodematch.eversmk1 1755.000 1646.000 1731.830 1847.000 0.70
## mutual 486.000 410.000 465.890 527.000 0.64
## gwesp.OTP.fixed.0.5 1775.406 1530.405 1690.474 1905.719 0.48
## gwdsp.OTP.fixed.0.5 13187.633 11220.527 12642.571 14646.853 0.44
##
## Goodness-of-fit for minimum geodesic distance
##
## obs min mean max MC p-value
## 1 2475 2294 2423.05 2622 0.44
## 2 10672 9223 10480.93 12074 0.74
## 3 31134 30852 36074.17 41709 0.02
## 4 50673 61190 65958.18 71501 0.00
## 5 42563 35578 41259.49 48248 0.70
## 6 18719 5072 9728.74 14627 0.00
## 7 4808 444 1402.11 2760 0.00
## 8 822 16 174.57 888 0.02
## 9 100 0 21.66 353 0.08
## 10 7 0 2.40 54 0.16
## 11 0 0 0.25 8 1.00
## 12 0 0 0.01 1 1.00
## Inf 12333 2079 6780.44 11879 0.00
##
## Goodness-of-fit for out-degree
##
## obs min mean max MC p-value
## 0 4 3 7.65 14 0.28
## 1 28 11 19.93 30 0.14
## 2 45 20 33.48 51 0.08
## 3 50 27 43.75 59 0.42
## 4 54 30 51.34 65 0.64
## 5 62 35 52.02 71 0.24
## 6 40 36 50.11 71 0.16
## 7 28 33 44.18 58 0.00
## 8 13 23 35.64 50 0.00
## 9 16 16 27.49 38 0.04
## 10 20 9 19.90 34 1.00
## 11 8 5 13.51 21 0.10
## 12 11 3 8.70 17 0.44
## 13 13 0 4.49 14 0.02
## 14 6 0 3.00 7 0.12
## 15 6 0 1.42 5 0.00
## 16 7 0 0.79 3 0.00
## 17 4 0 0.30 2 0.00
## 18 3 0 0.18 2 0.00
## 19 0 0 0.06 1 1.00
## 20 0 0 0.06 1 1.00
##
## Goodness-of-fit for in-degree
##
## obs min mean max MC p-value
## 0 13 2 7.48 13 0.08
## 1 34 10 19.25 30 0.00
## 2 37 26 34.83 46 0.82
## 3 48 31 44.19 61 0.54
## 4 37 32 50.78 67 0.06
## 5 47 33 51.87 67 0.50
## 6 42 37 49.08 69 0.32
## 7 39 30 43.71 58 0.52
## 8 35 19 36.73 51 0.80
## 9 21 14 27.81 38 0.28
## 10 12 12 20.01 34 0.02
## 11 19 6 13.62 24 0.20
## 12 4 2 8.07 18 0.18
## 13 7 0 4.96 14 0.42
## 14 6 0 3.04 11 0.24
## 15 3 0 1.35 6 0.38
## 16 4 0 0.68 4 0.02
## 17 3 0 0.33 3 0.02
## 18 3 0 0.11 1 0.00
## 19 2 0 0.04 1 0.00
## 20 1 0 0.03 1 0.06
## 21 0 0 0.02 1 1.00
## 22 1 0 0.01 1 0.02
##
## Goodness-of-fit for edgewise shared partner
##
## obs min mean max MC p-value
## .OTP0 1032 932 1010.98 1087 0.60
## .OTP1 755 690 800.77 908 0.34
## .OTP2 352 318 394.89 464 0.10
## .OTP3 202 112 152.00 201 0.00
## .OTP4 79 24 47.71 81 0.02
## .OTP5 36 2 12.00 24 0.00
## .OTP6 14 0 3.72 18 0.06
## .OTP7 4 0 0.91 6 0.10
## .OTP8 1 0 0.06 2 0.10
## .OTP9 0 0 0.01 1 1.00
# Graficando estadísticas GOF
plot(ans_gof)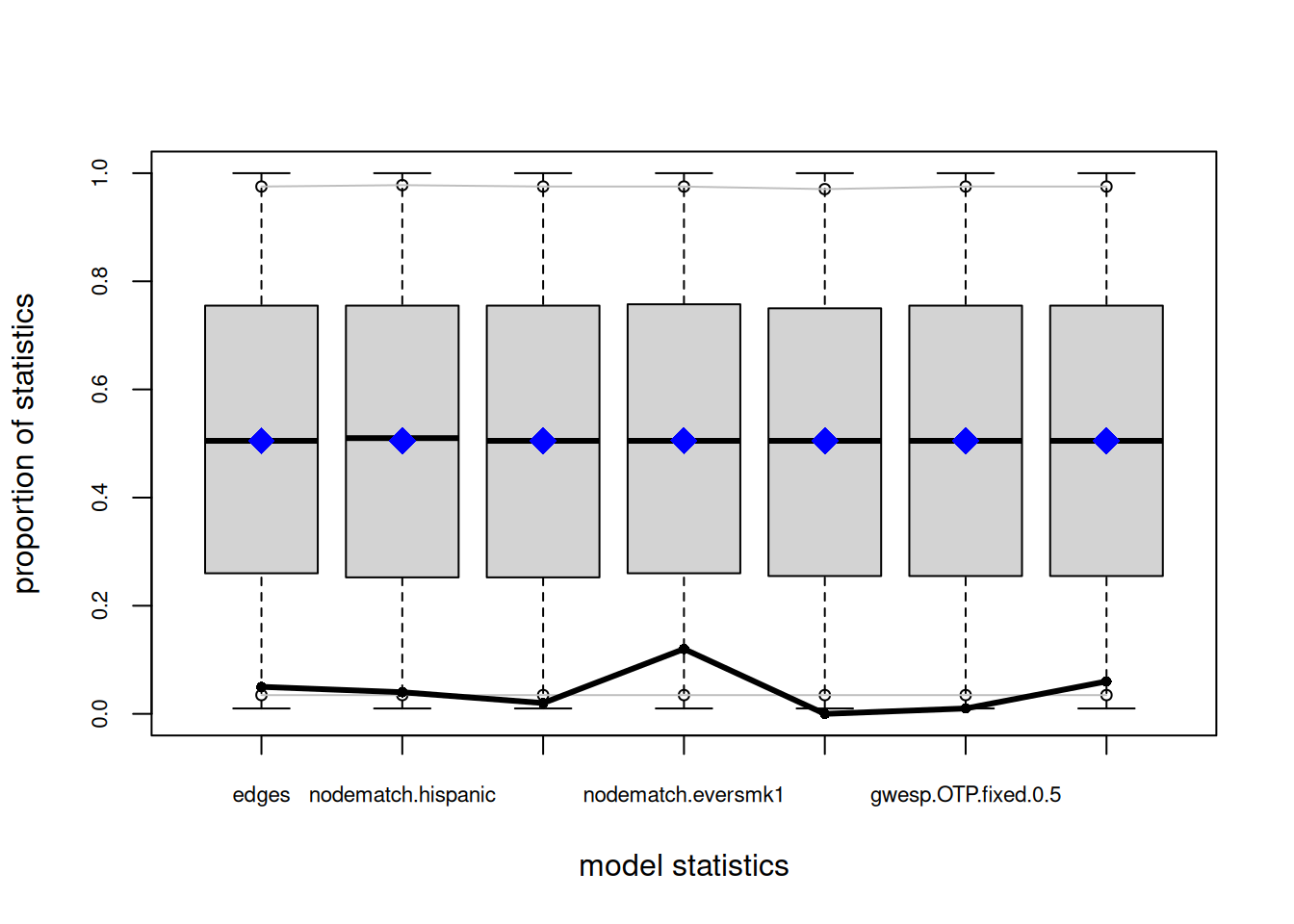
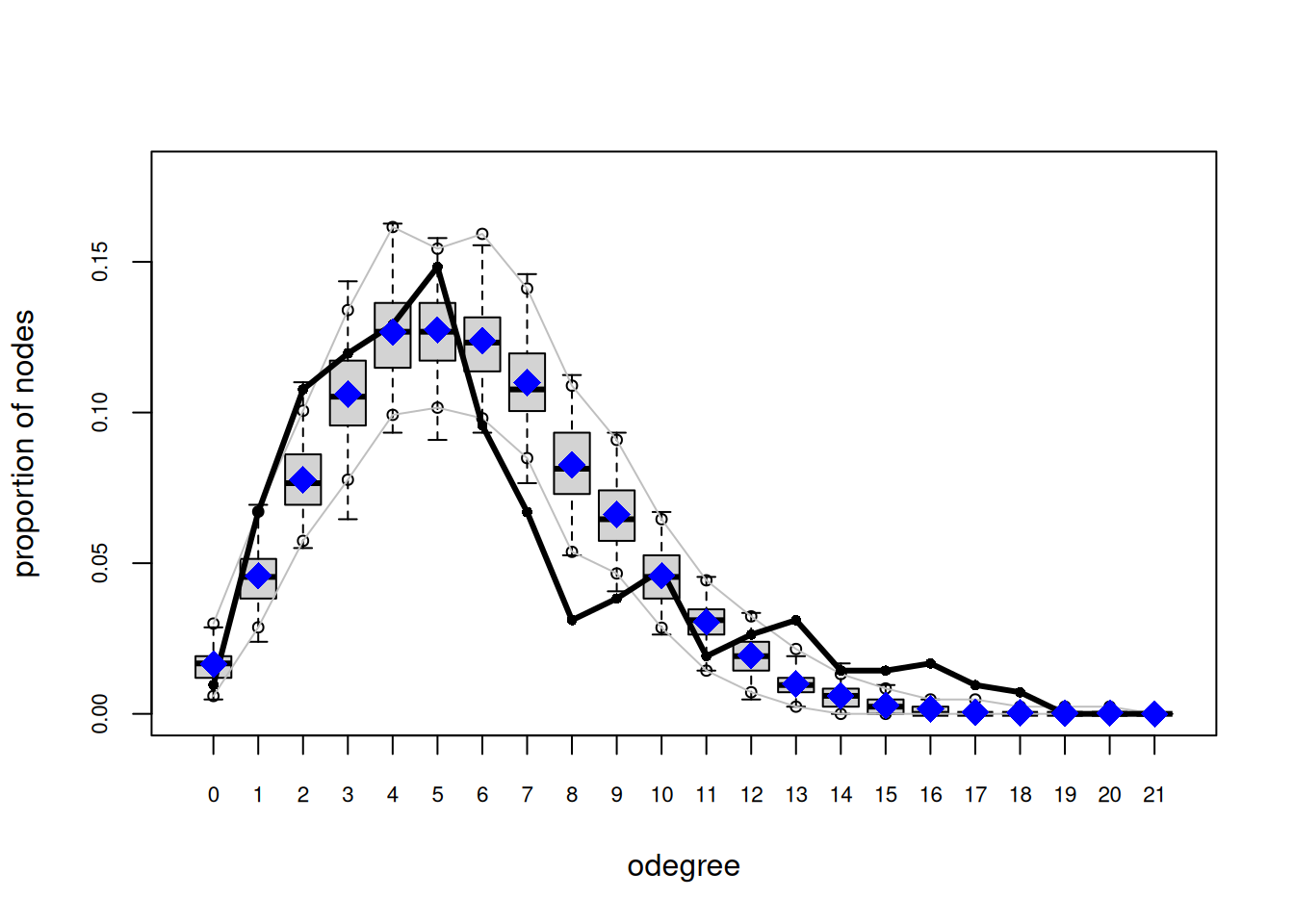
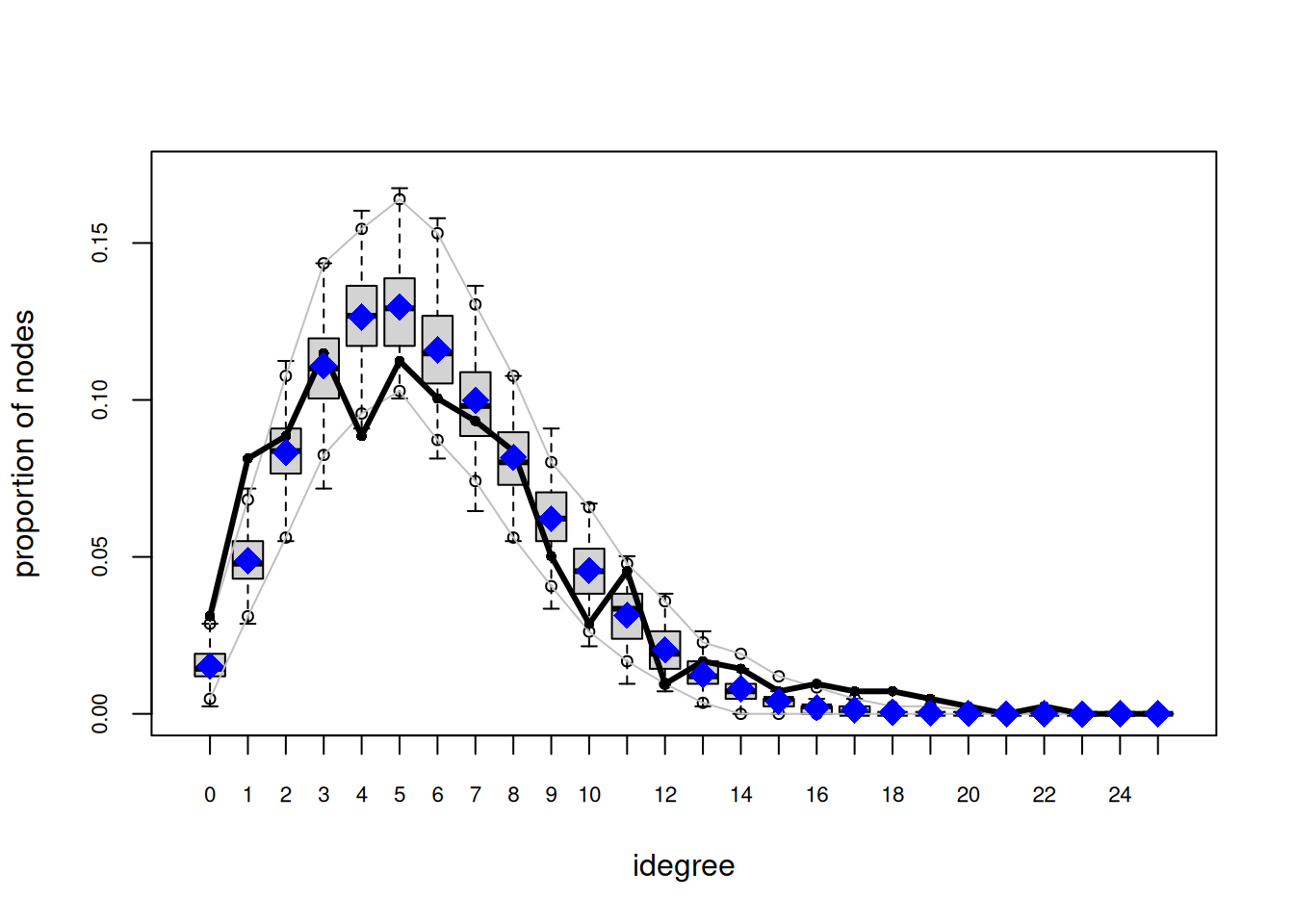
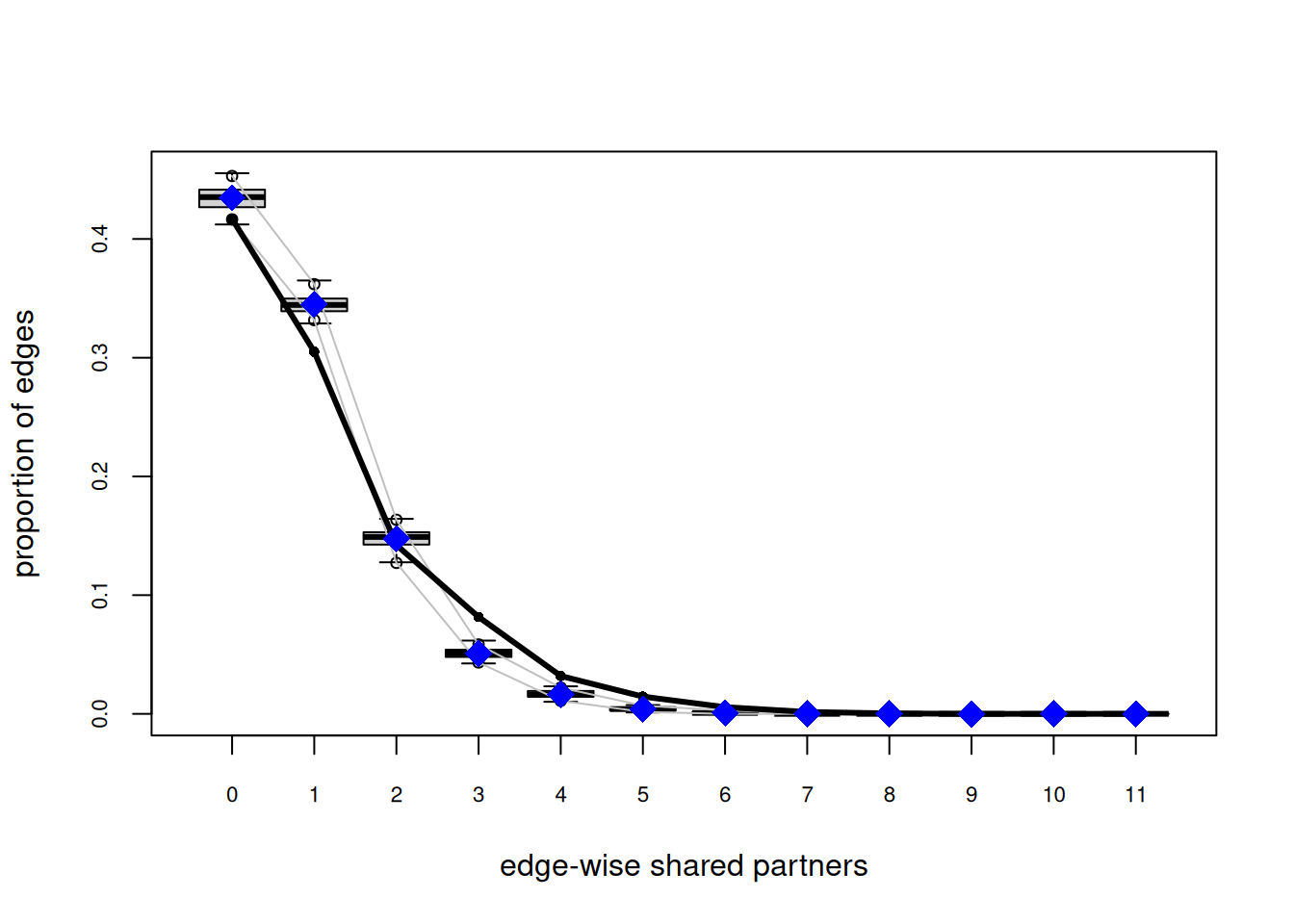
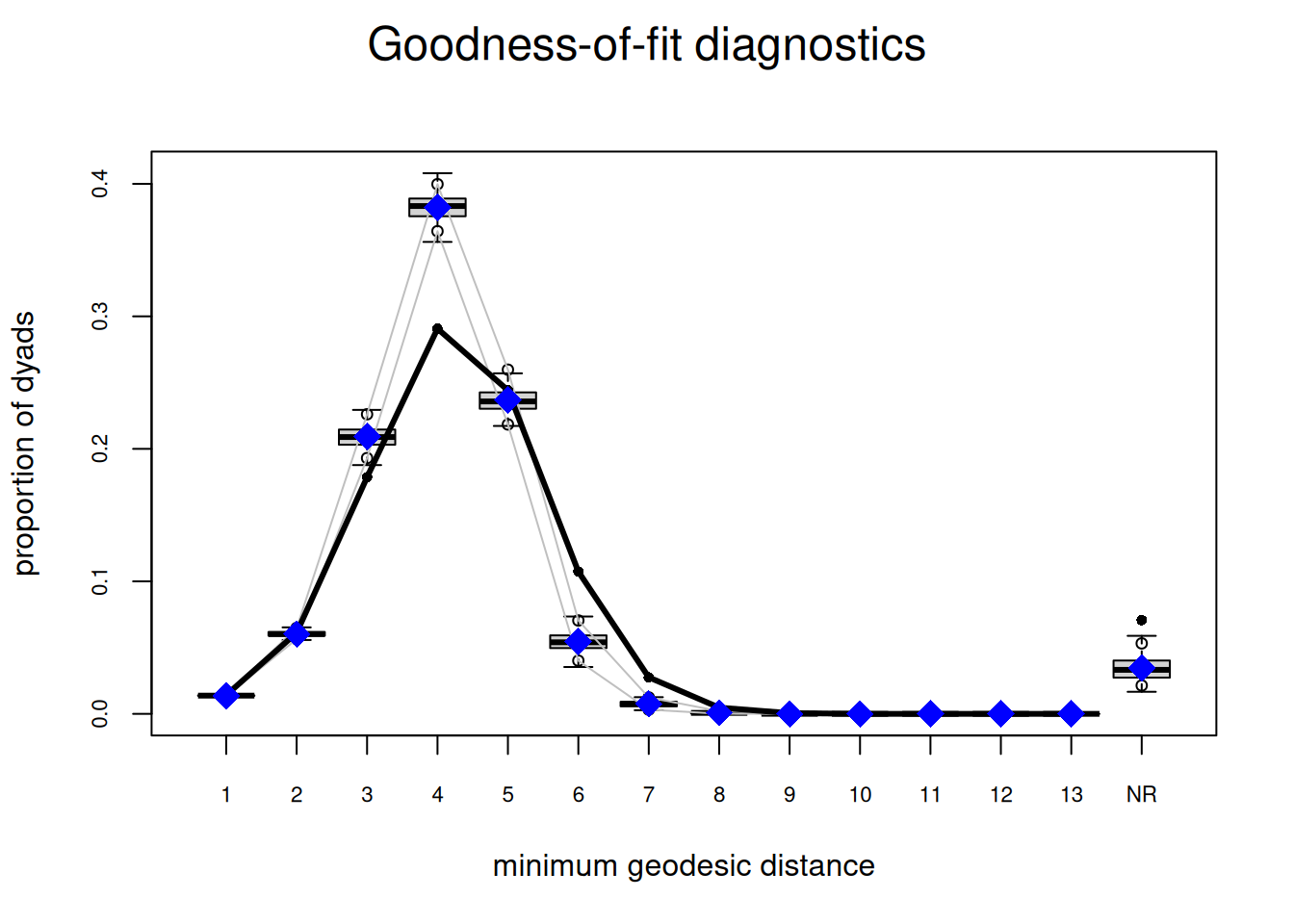
Prueba la siguiente configuración en su lugar
ans0_bis <- ergm(
network_111 ~
edges +
nodematch("hispanic") +
nodematch("female1") +
mutual +
esp(0:3) +
idegree(0:10)
,
constraints = ~bd(maxout = 19),
control = control.ergm(
seed = 1,
MCMLE.maxit = 15,
parallel = 4,
CD.maxit = 15,
MCMC.samplesize = 2048*4,
MCMC.burnin = 30000,
MCMC.interval = 2048*4
)
)Aumentar el tamaño de muestra, para que las curvas sean más suaves, intervalos más largos (adelgazamiento), lo que reduce la autocorrelación, y un quemado más grande. Todo esto junto para mejorar la estadística de prueba de Gelman. También agregamos idegree del 0 al 10, y esp del 0 al 3 para coincidir explícitamente con esas estadísticas en nuestro modelo.
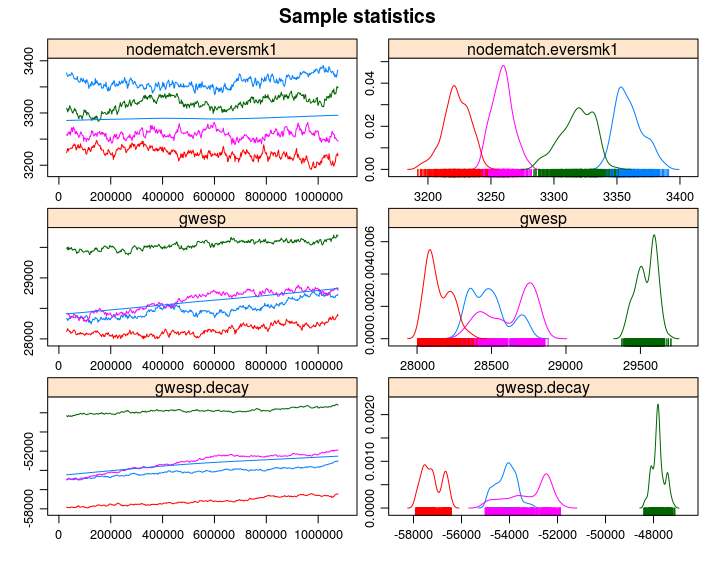
Para más sobre este tema, recomiendo revisar capítulo 1 y capítulo 6 del Manual de MCMC (Brooks et al. 2011). Ambos capítulos están disponibles para descarga gratuita desde el sitio web del libro.
Para GOF echa un vistazo a la sección 6 del tutorial ERGM 2016 Sunbelt, y para una revisión más técnica, puedes echar un vistazo a (David R. Hunter, Goodreau, and Handcock 2008).
Una de las partes más críticas del modelado estadístico es interpretar los resultados, si no la más importante. En el caso de ERGMs, un aspecto clave se basa en estadísticas de cambio. Supón que nos gustaría saber qué tan probable es que el vínculo y_{ij} ocurra, dada el resto de la red. Podemos calcular tales probabilidades usando lo que la literatura a veces describe como el muestreador de Gibbs.
En particular, las log-odds del vínculo ij ocurriendo condicional en el resto de la red pueden escribirse como:
\begin{equation} \text{logit}\left({\Pr{y_{ij} = 1|y_{-ij}}}\right) = {\theta}^\mathbf{t}\Delta\delta\left(y_{ij}:0\to 1\right), \end{equation}
con \delta\left(y_{ij}:0\to 1\right)\equiv s\left(\mathbf{y}\right)_{\text{ij}}^+ - s\left(\mathbf{y}\right)_{\text{ij}}^- como el vector de estadísticas de cambio, en otras palabras, la diferencia entre las estadísticas suficientes cuando y_{ij}=1 y su valor cuando y_{ij} = 0. Para mostrar esto, escribimos lo siguiente:
\begin{align*} \Pr{y_{ij} = 1|y_{-ij}} & = % \frac{\Pr{y_{ij} = 1, y_{-ij}}}{% {\mathbb{P}_{+}\left(y_{ij} = 1, y_{-ij}\right) } \Pr{y_{ij} = 0, y_{-ij}}} \\ & = \frac{\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^+_{ij}\right\}}{% \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^+_{ij}\right\} + \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^-_{ij}\right\}} \end{align*}
Aplicando la función logit a la ecuación anterior, obtenemos:
\begin{align*} & = \text{log}\left\{\frac{\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^+_{ij}\right\}}{% \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^+_{ij}\right\} + % \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^-_{ij}\right\}}\right\} - % \text{log}\left\{ % \frac{\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^-_{ij}\right\}}{% \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^+_{ij}\right\} + \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^-_{ij}\right\}}% \right\} \\ & = \text{log}\left\{\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^+_{ij}\right\}\right\} - \text{log}\left\{\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)^-_{ij}\right\}\right\} \\ & = {\theta}^\mathbf{t}\left(s\left(\mathbf{y}\right)^+_{ij} - s\left(\mathbf{y}\right)^-_{ij}\right) \\ & = {\theta}^\mathbf{t}\Delta\delta\left(y_{ij}:0\to 1\right) \end{align*} Por lo tanto, la probabilidad condicional de que el vínculo y_{ij} ocurra puede escribirse como:
\begin{equation} {\mathbb{P}_{=}\left(y_{ij} = 1|y_{-ij}\right) } \frac{1}{1 + \text{exp}\left\{-{\theta}^\mathbf{t}\Delta\delta\left(y_{ij}:0\to 1\right)\right\}} \end{equation}
es decir, una probabilidad logística.
El desafío de analizar redes es su naturaleza interdependiente. No obstante, en ausencia de tal interdependencia, los ERGMs son equivalentes a regresión logística. Conceptualmente, si todas las estadísticas incluidas en el modelo no involucran dos o más díadas, entonces el modelo pierde complejidad.
Matemáticamente, para ver esto, es suficiente mostrar que la probabilidad ERGM puede escribirse como el producto de las probabilidades de cada díada.
{\mathbb{P}_{=}\left(\mathbf{y} | \theta\right) } \frac{\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)\right\}}{\sum_{y}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)\right\}} = \frac{\prod_{ij}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)_{ij}\right\}}{\sum_{y}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)\right\}}
Donde s\left(\right)_{ij} es una funcion tal que s\left(\mathbf{y}\right) = \sum_{ij}{s\left(\mathbf{y}\right)_{ij}}. Ahora, debemos lidear con la constante normalizadora. Para esto, necesitamos usar el hecho de que podemos intercambiar la suma y el producto. En el caso del modelo de díada independiente, la constante normalizadora puede escribirse como:
\sum_{y\in\mathcal{Y}}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)\right\} = % \sum_{y\in\mathcal{Y}}\prod_{ij}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)_{ij}\right\} = % \prod_{ij}\sum_{y\in\{0,1\}}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}_{ij}\right)\right\}
Lo anterior solo es posible si las díadas son idependientes. De lo contrario, tal arreglo no es factible. Ahora, podemos re escribir la ecuación como sigue:
\prod_{ij}\left[\frac{% \text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)_{ij}\right\}% }{% \sum_{y'\in\{0,1\}}\text{exp}\left\{{\theta}^\mathbf{t}s\left(\mathbf{y}\right)_{ij}\right\}% }\right] = % \prod_{ij}\left[\frac{1}{% 1 + \text{exp}\left\{-{\theta}^\mathbf{t}\Delta\delta\left(y_{ij}:0\to 1\right)\right\}}% \right]
Donde la última igualdad está dada al dividir cada elemento de la multiplicación por su numerador correspondiente. Dado que al menos uno de los dos términos de la suma \sum_{y'\in\{0,1\}} es igual al observado en la red, uno de ellos equivale a uno y el otro a una función del estadístico de cámbio.
Relacionado con esto, los ERGMs bloque-diagonales pueden estimarse como modelos independientes, uno por bloque. Para ver más sobre esto, lee (SNIJDERS 2010). De manera similar, dado que la independencia depende–juego de palabras intencionado–de particionar la función objetivo, como señala Snijders, las funciones no lineales hacen que el modelo sea dependiente, ej., s\left(\mathbf{y}\right) = \sqrt{\sum_{ij}y_{ij}}, la raíz cuadrada del conteo de enlaces ya no es un grafo de Bernoulli.
Puedes descargar el archivo 03.rda desde este enlace.↩︎
Sí, las clases tienen el mismo nombre que los paquetes.↩︎
El sitio web wiki de statnet tiene un ejemplo muy agradable de gráficos de diagnóstico MCMC (muy) malos y buenos aquí.↩︎